El diseño de ediciones para secuencias biológicas es un campo donde hay mucho márgen de mejora, y una de las herramientas más prometedoras en este ámbito es la que nos toca hoy. Ledidi es un enfoque innovador que invierte la forma tradicional de pensar sobre los modelos de aprendizaje automático, manteniendo constante el modelo y actualizando los datos en su lugar. Esto permite a Ledidi convertir cualquier modelo de aprendizaje automático capacitado en un editor de secuencias biológicas, independientemente de su propósito original.
Uno de los desafíos más significativos al diseñar ediciones para secuencias categóricas, como las secuencias de ADN, es que las entradas suelen estar codificadas de un solo disparo, mientras que los gradientes son continuos. Esto puede producir resultados no biológicamente significativos, como valores de 1,2 de A y -0,3 de G, que no pueden ser diseñados en una secuencia real. Ledidi resuelve este desafío formulando el diseño de edición como un problema de optimización sobre una matriz de pesos continuos, lo que permite aplicar los gradientes directamente a la secuencia.
Cómo funciona Ledidi a la hora de secuenciar proteínas
El proceso de Ledidi comienza con la selección de un modelo de aprendizaje automático basado en secuencias, una secuencia inicial y un resultado deseado del modelo. Luego, se pasa estos tres elementos a Ledidi para diseñar la edición. Ledidi utiliza una distribución de Gumbel-softmax para muestrear secuencias codificadas de un solo disparo, una posición a la vez, y actualiza los pesos de la matriz para preferir ciertos caracteres. Esto se repite en cada iteración, y el resultado es una secuencia editada que se pasa a través del modelo preentrenado congelado para comparar la salida con la salida deseada.
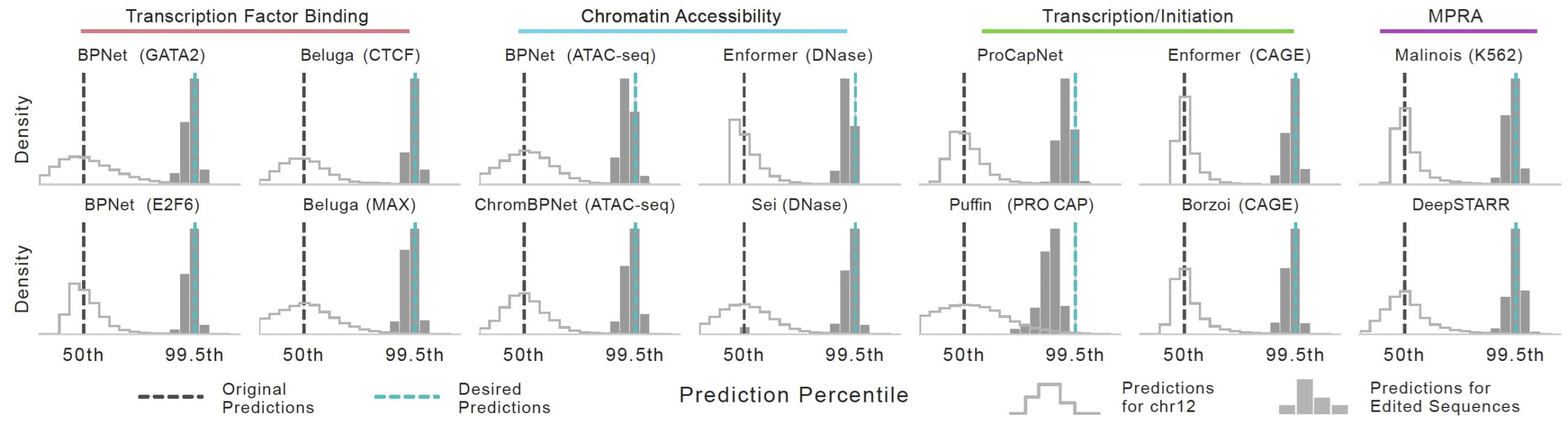
La pérdida total se deriva de la pérdida de salida y de una pérdida de entrada que es el recuento de ediciones propuestas. Esto limita el número de ediciones que Ledidi propondrá, y el gradiente se calcula a partir de esta pérdida para actualizar los pesos de la matriz. Ledidi puede diseñar ediciones que conviertan una secuencia que no exhiba alguna forma de actividad en una que exhiba una actividad fuerte, y puede aplicarse a modelos de ARN o proteínas, o a cualquier modelo con una secuencia de entradas categóricas.
Cómo empezar a utilizar Ledidi
La instalación de Ledidi es sencilla, y puede realizarse mediante pip: pip install ledidi. El uso de Ledidi requiere un modelo de aprendizaje automático basado en secuencias, una secuencia inicial y un resultado deseado del modelo. Por ejemplo, puede importar un modelo BPNet y utilizar Ledidi para diseñar ediciones que conviertan una secuencia que no exhiba actividad en una que exhiba una actividad fuerte.
El código para utilizar Ledidi es el siguiente: import torch; from bpnetlite.bpnet import ControlWrapper; from bpnetlite.bpnet import CountWrapper; from ledidi import ledidi; X = .. tu secuencia ..; modelo = torch.load("GATA2.torch", map_location=torch.device('cuda')); modelo = CountWrapper(ControlWrapper(modelo)); y_bar = torch.zeros(1, dtype=torch.float32) + 4.5; X_hat = ledidi(modelo, X, y_bar). Esto ejecutará Ledidi y devolverá un lote de secuencias editadas que pueden ser utilizadas para diseñar ediciones que conviertan una secuencia que no exhiba actividad en una que exhiba una actividad fuerte.
Los registros de Ledidi pueden ser útiles para entender el proceso de diseño de ediciones. Por ejemplo, puede ver la pérdida de entrada y salida, y la pérdida total al combinarlas con el peso de mezcla. Los registros pueden verse así: Iteración 100, pérdida de entrada: 35,75, pérdida de salida: 16, pérdida total: 0,18. Esto significa que en la iteración 100, se propuso un promedio de 35,75 ediciones por secuencia, y que la mejor iteración en convergencia solo tuvo 28,19 ediciones por secuencia en promedio.
Puedes echar un vistazo a su repositorio aquí. Si te interesa este tema, te dejo el enlace a su guía oficial donde hay, además, tutoriales de uso.